服务介绍
SNP分型
单核苷酸多态性(Single Nucleotide Polymorphisms,SNPs),即指由于单个核苷酸碱基的改变而导致的核酸序列的多态性。在不同个体的同一条染色体或同一位点的核苷酸序列中,绝大多数核苷酸序列一致而只有一个碱基不同的现象,这就是SNP。由于SNP在人类基因组中数量较多,发生频率较高,因此被认为是继微卫星之后的新一代遗传学标记,在医学遗传学、药物遗传学、疾病遗传学、疾病诊断学、以及人类进化等研究领域都有着很高的研究价值和应用前景。
本公司在SNP位点分析和研究方面积累了丰富的经验,拥有开展SNP研究的先进仪器设备,具有自己独到的研究策略和方法,并且已经建立大样本量人群的SNP基础数据库,能通过这些资源的有效挖掘,更好地帮助客户开展SNP方面的研究工作,包括SNP的功能学研究。
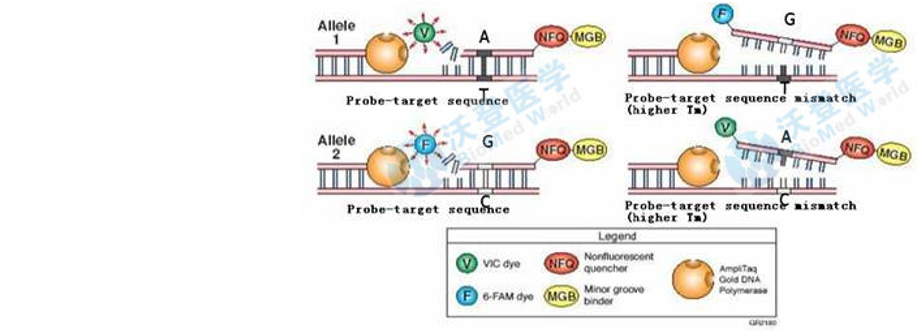
SNP分型原理图
服务内容
1.质谱分析法(mass spectrometry)
该方法利用质谱分析对质量的灵敏度特别高的特点,很容易将仅含有一个不同碱基的两段基因序列区别开的特点,使用质谱直接或间接检测等位基因特异性延伸区的反应产物,推导出SNP。单碱基延伸或其修饰形式与基质辅助激光吸收/离子飞行时间(MALDI-TOF)质谱结合已被广泛的应用,并已被美国公司(Sequenom)发展为商业性产品。我公司提供的SNP检测技术服务,就是基于这样的方法。
2.DNA芯片技术(DNA chip)
将已知序列的寡核苷酸DNA排列在1块集成电路板上,将荧光标记的正常DNA和突变DNA分别与2块DNA芯片杂交,由于至少存在1个碱基的差异,正常和突变的DNA将会得到不同的杂交图谱,经过共聚集显微镜检测两种DNA分子产生的荧光信号,确定是否存在突变。
3.限制性片段长度多态性分析法(RFLP)
RFLP技术利用限制性核酸内切酶识别并剪切特定DNA序列的能力,检测基因片段上限制性核酸内切酶识别位点处是否存在核苷酸突变。应用限制性内切酶对两个等位基因识别的差异,产生不同大小的切割片段,通过电泳迁移率的不同来判定是否存在SNP。
4.实时荧光PCR分析法
使用针对核酸中不同多态性序列的荧光Taqman探针,它们在PCR扩增中被水解释放荧光信号,只有与靶序列完全匹配的探针才能被相应的切割。不同探针标记不同的荧光报道信号。利用多通道荧光PCR仪检测结果,可以便捷判断核酸多态性。
5.单链构象多态性分析法(SSCP)
在非变性聚丙烯酰胺凝胶上,短的单链DNA和RNA分子依其大碱基序列不同而形成不同构象,一个碱基的改变将影响其构象而导致其在凝胶上的移动速度改变。
6.DNA测序法(DNA sequencing)
双脱氧终止法,引物用四种不同颜色的荧光标记,使每个样品的四个测序反应可在一个反应管和一个泳道内进行。
服务说明
1.样品纯度:OD 260/280值应在1.8~2.0 之间;RNA 应该去除干净;
2.样品浓度:低浓度不低于50 ng/µL;
3.样品总量:每个样品总量不少于15 µg;
4.样品溶剂:要溶解在H2O或TE (pH 8.0)中;
5.样品运输: DNA低温运输(-20℃);且在运输过程中请用parafilm将管口密封好,以防出现污染;
6.若另有疑问欢迎向本公司咨询,我们将竭诚为您服务
质量保证
1.准确可靠的测序结果;
2.专业权威的分析报告;
3.真实的原始实验数据;
4.详细的实验步骤。


